近日,由中国科学院微生物研究所国家微生物科学数据中心开发的“全球宏基因组数据库” (global catalogue of metagenome, gcMeta , https://gcmeta.wdcm.org/), 在《Nucleic Acids Research》数据库专刊发表。该数据库整合了超 270万MAGs,涵盖 10.4万余个样本、50个特定生物群落,不仅填补了跨生态系统的微生物比较分析的空白,更为全球科研人员提供了标准化、可直接用于人工智能(AI)研究的微生物功能数据资源,开启了微生物 “序列发现” 到 “功能利用” 的高效转化通道。

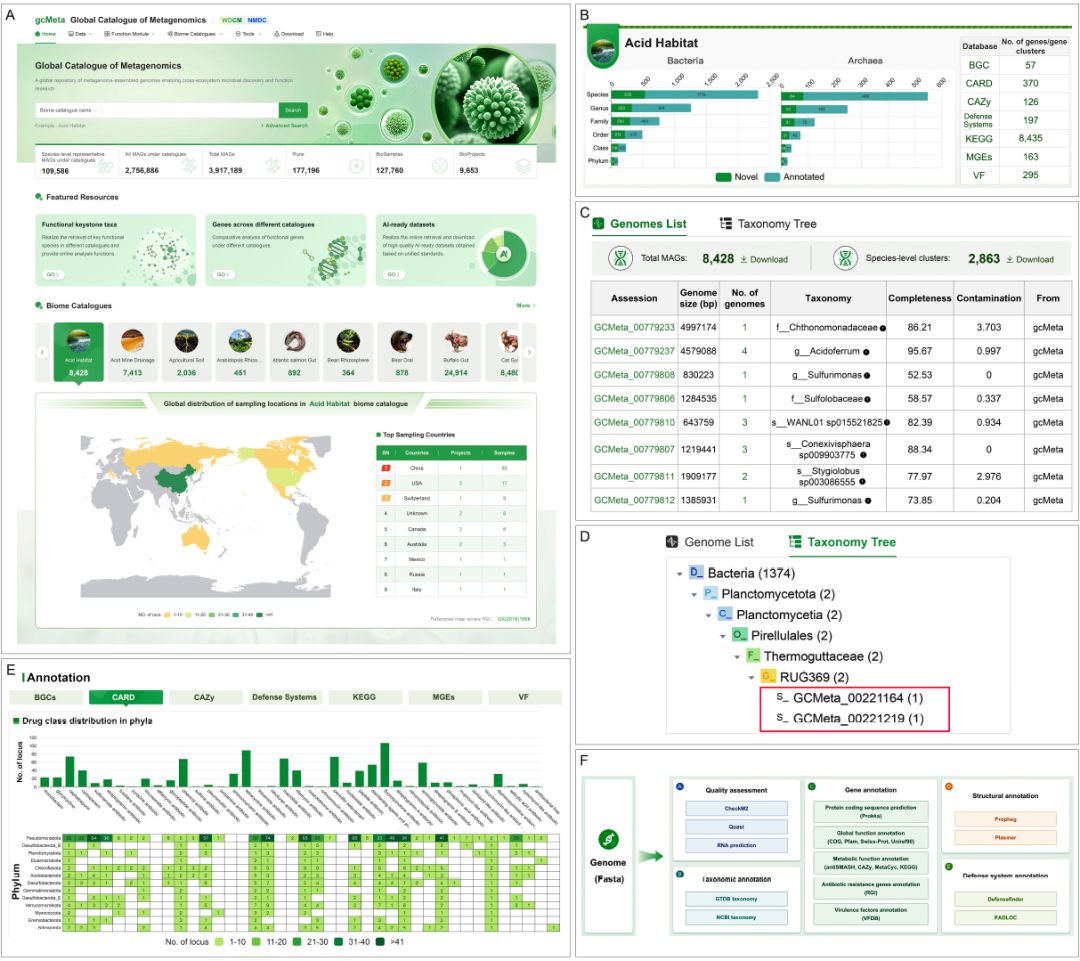
图2. gcMeta 网页。(A)gcMeta 主页。(B)特定生物群落宏基因组组装基因组(MAG)目录概述。(C)每个目录中的基因组列表。(D)分类树可按目标类群检索基因组。(E)每个目录中的注释部分。(F)在线数据分析流程。
从 “物种识别” 到 “功能预测” 再到“比较分析”:六大核心模块覆盖微生物研究全链条
gcMeta集成了标准化比较分析框架、分析工具与可视化界面的科研平台,可满足从基础基因组分析到深度功能挖掘,再到跨生态系统比较分析的多样化需求。
1. 270万MAGs+50个特色基因组目录:构建全球最全面的宏基因组组装基因组资源库
gcMeta 2025的海量、跨生态系统的高质量数据资源,彻底打破了传统数据库 “单一环境聚焦” 的局限。研究团队通过 “公共数据整合+从头组装” 双路径,系统收集了NCBI SRA及国家微生物科学数据中心NMDC等数据库中符合标准的宏基因组原始数据,并结合 MGnify、IMG/M、SPIRE、mOTUs 四大国际平台的MAGs资源,最终形成覆盖人类、动物、植物、海洋、淡水、极端环境等12大类群(各类极端环境、人与动物宿主、植物根际等)、50个特色生物群落的MAGs目录。宏基因组测序数据。数据经过统一的质量控制及注释流程,进行物种及功能注释,网站提供不同分类等级下已注释物种与新物种的列表及所有基因组的注释信息的浏览及下载,极大方面了从事特定类群微生物组及比较微生物组研究的工作。
2. 跨生态系统比较分析:揭示微生物 “环境适应密码”
“功能模块(Function Module)” 能够对MAGs目录中的物种组成和功能多样性进行比较分析。“分类丰度” 部分支持在 GTDB 或 NCBI 分类体系下进行检索,提供从门到属的筛选选项,以及直接的分类单元搜索功能。功能比较涵盖七个主要类别:抗生素耐药基因(ARGs)、毒力因子(VFs)、KEGG 代谢通路、生物合成基因簇(BGCs)、碳水化合物活性酶(CAZymes)、可移动遗传元件(MGEs)和防御系统。用户可以按亚组(例如,抗生素耐药基因的药物类别、通路模块)比较功能基因的分布,并对特定基因或基因簇进行分类检索。
3. 功能关键类群识别:锁定生态系统 “核心玩家”
通过整合 “基因组功能特征” 与 “物种共现网络”,平台可定位各生态系统中的 “功能关键类群”(图3C),并将其与生物地球化学循环(如氮/硫代谢)和环境适应(如耐盐性/重金属抗性)联系起来。此模块还可以点击获取关键物种的核心功能基因携带及其在不同生态系统中的分布信息。
4. 特色基因集:支撑多生态系统功能基因比较与新基因挖掘
此模块支持对非冗余基因集(包括功能性酶和新基因)及其在各类数据库中的注释结果进行统计比较(图3D)。用户可以通过 “条件检索” 功能生成基因列表,或进行个性化搜索(图3E),以比较不同目录中的基因分布。“流行基因” 可跨生态系统挖掘核心物种功能,支持比较不同目录中共有基因与特有基因,并提供可下载的基因列表(图3F)。这为微生物生态适应研究提供了系统性见解,并有助于发现新的功能元件。
5. AI-ready数据集:直接支撑机器学习模型训练
针对当前微生物 AI 研究中 “数据格式不统一、质量参差不齐” 的痛点,gcMeta平台专门构建了78个标准化 AI-ready 数据集,涵盖三大类:1)生物地球化学循环代谢酶:如氮循环中的固氮酶、硫循环中的脱硫酶;2)核心功能酶:包括 α-淀粉酶、β-葡萄糖苷酶等工业常用酶;3)防御系统组件:涵盖13类已知微生物防御系统(如 CRISPR-Cas、限制修饰系统)。这些数据集可直接用于蛋白质语言大模型的训练与微调,为微生物 AI 研究提供 “即用型” 数据支持。
6. 在线MAG分析流程:零基础完成基因组分析
gcMeta 提供了一个用于宏基因组组装基因组(MAG)分析的交互式分析工具(图 2F)。工具支持基因组质量评估、分类注释、基因注释、结构注释以及防御系统注释。用户可以通过 “program selection” 模块自定义分析,从而实现根据特定需求定制灵活的分析流程。
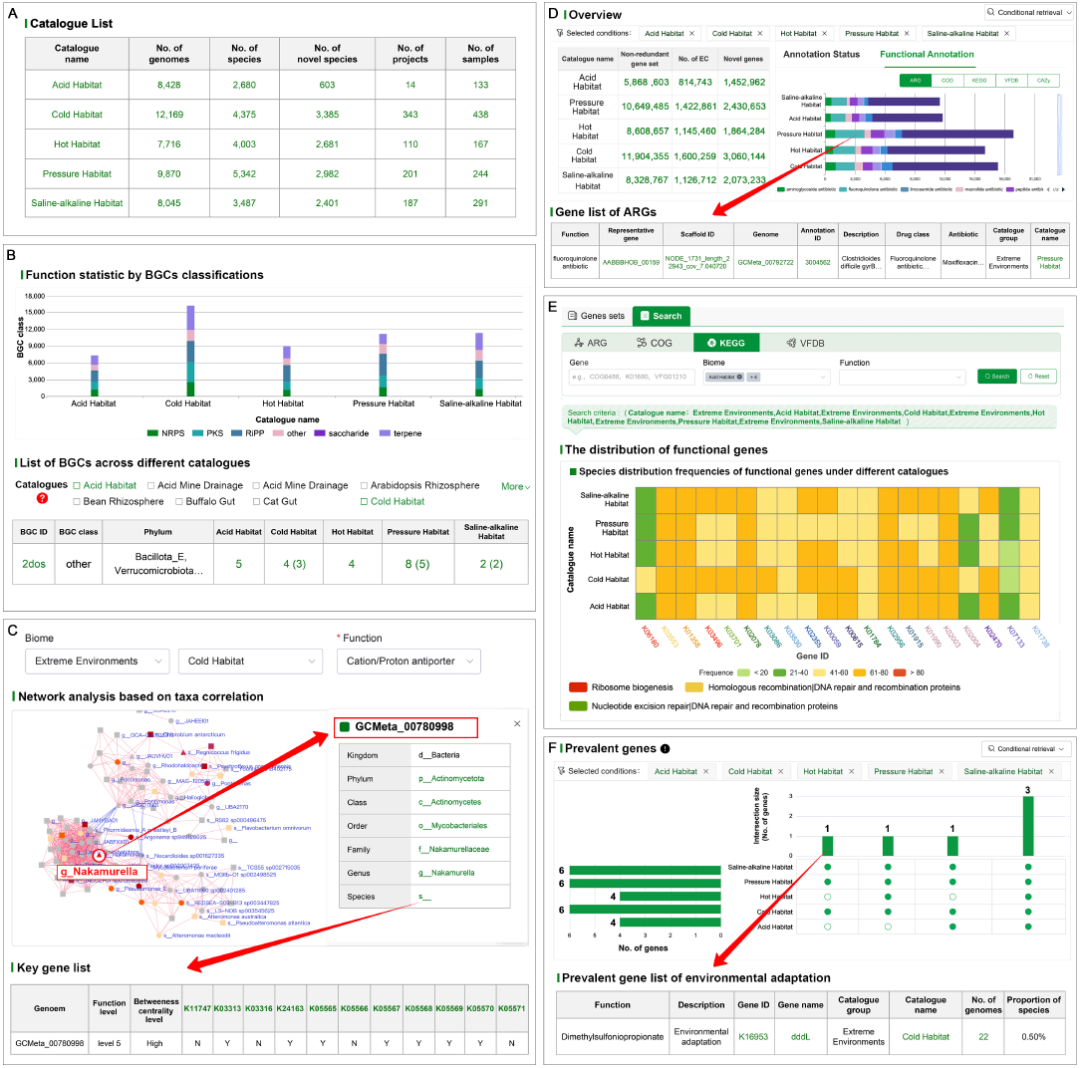
图 3. 不同 MAGs 目录的比较分析。(A)“目录列表”,包括基因组、物种和样本统计数据。(B)“功能模块” 下 BGC 注释的统计比较。(C)与极端冷生境中阳离子/质子反向转运蛋白相关的关键类群。(D)非冗余基因集的统计比较,突出显示新基因和功能基因的分布。(E)跨生境基因的在线检索和比较统计。(F)核心物种中流行基因的功能分析。
gcMeta 2025不仅为微生物研究提供了跨生态系统的微生物组成及功能比较分析平台,更搭建了 “基础研究-技术开发-产业应用” 的桥梁,有望加速微生物资源的挖掘与利用,为解决生态环境、人类健康、工业生产等领域的关键问题提供新方案。



